without haste but without rest
09. Clustering - dbscan, spectal 본문
0. 개요
- dbscan은 k-means 보다는 connectiviy 하고
- spectral 보다는 compactness 하다.
1. dbscan
- 코어 데이터에서 반지름인 epsilon 을 기준으로 해당 원 안에 들어오는 데이터들을 군집으로 묶어 나간다.
- 묶인 데이터가 가장 바깥에 위치하면 해당 데이터는 border 데이터, 어디에도 속하지 않는다면 noise 데이터
- moons 데이터와 같은 데이터에서 좋은 성능을 보인다. -> 클러스터 개수가 적은 데이터
- 클러스터 개수가 많은 데이터에서는 좋은 성능을 내지 못한다.
- k-means는 moons 데이터에와 같은 자료형에서 좋은 성능을 못낸다.
- 경우에 따라서 수치 범위를 보고 표준화를진행해주면 k-means 도 더 좋은 성능을 낼 수 있다.
- https://en.wikipedia.org/wiki/DBSCAN
DBSCAN - Wikipedia
From Wikipedia, the free encyclopedia Jump to navigation Jump to search A density-based data clustering algorithm Density-based spatial clustering of applications with noise (DBSCAN) is a data clustering algorithm proposed by Martin Ester, Hans-Peter Krieg
en.wikipedia.org
2. spectral
- 앞서 다룬 어떤 모델보다도 성능이 좋다.
- 그러나 계산량이 많고,
- 초기 클러스터 개수를 정하기가 어렵다.
- 강의 scope에서 벗어나므로 이론적 학습은 진행하지 않고 모델을 돌려보기만 한다.
1. dbscan
1-1 샘플 데이터 생성
"""
dbscan, spectral 은
connectivity 에 중점을 둔 알고리즘이다.
클러스터링 알고리즘은 보통 compactness vs connectivity 두 가지에 초점을 맞춘다.
전 주에 다룬 k-means, hierarchical = compactness
"""
import pandas as pd
import seaborn as sns
sns.set_context("paper", font_scale=1.5)
sns.set_style("white")
## 클러스터링 알고리즘 학습을 위한 가상 자료 생성 ##
###########################################################################
from sklearn import datasets
def make_blobs():
# build blobs for demonstration
n_samples = 1500
blobs = datasets.make_blobs(n_samples = n_samples,
centers = 5,
cluster_std = [3.0, 0.9, 1.9, 1.9, 1.3],
random_state = 51)
# create a Pandas dataframe for the data
df = pd.DataFrame(blobs[0], columns=['Feature_1', 'Feature_2'])
df.index.name = 'record'
return df
df = make_blobs()
print(df.head(10))
# plot scatter of blob set
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
data = df, fit_reg=False)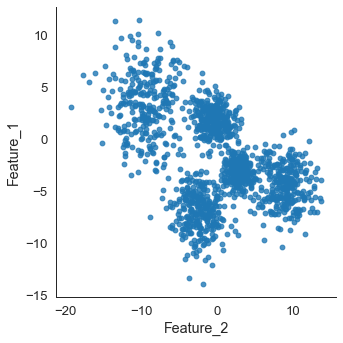
해당 데이터들을 군집화할 수 있는 비지도 학습 알고리즘 중에서 dbscan, spectral 알고리즘을 사용한다.
1-2. dbscan
from sklearn.cluster import DBSCAN
# eps: epsilon
# min_sample: minPt
dbscan = DBSCAN(eps = 0.5, min_samples = 5, metric = 'euclidean')
# fit to input data
dbscan.fit(df)
# get cluster assignments
df['DBSCAN Cluster Labels'] = dbscan.labels_
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
hue = "DBSCAN Cluster Labels", data = df, fit_reg = False)
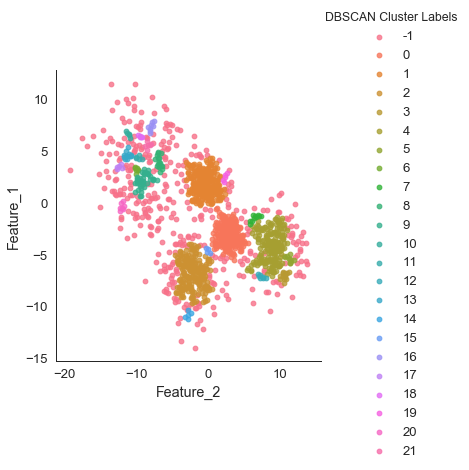
dbscan 결과 군집화가 영 좋지 못한 결과를 보인다. dbscan은 위와 같이 군집 개수가 많은 경우에서는 사용하기가 어렵다.
위에서 -1 군집이 노이즈 데이터이다.
2. moons data
2-1 데이터 생성
###########################################################################
## moons 자료 생성 ##
###########################################################################
def make_moons():
moons = datasets.make_moons(n_samples = 200, noise = 0.05, random_state = 0)
df = pd.DataFrame(moons[0], columns = ['Feature_1', 'Feature_2'])
df.index.name = 'record'
return df
moons = make_moons()
print(moons.head())
# plot scatter of blob set
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
data = moons, fit_reg = False)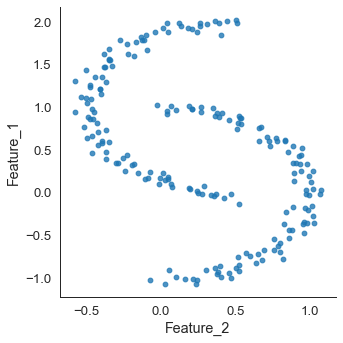
위 데이터에서 x 축과 y 축의 범위가 다른 것을 볼 수 있다. 따라서 표준화를 진행한다.
2-2 표준화
## 표준화
from sklearn.preprocessing import StandardScaler
# Rescale the data to zero mean and unit variance
scaler = StandardScaler()
scaler.fit(moons)
moons_scaled = scaler.transform(moons)
moons_scaled = pd.DataFrame(moons_scaled, columns = ['Feature_1', 'Feature_2'])
print(moons_scaled)
# plot scatter of moon set
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
data = moons_scaled, fit_reg = False)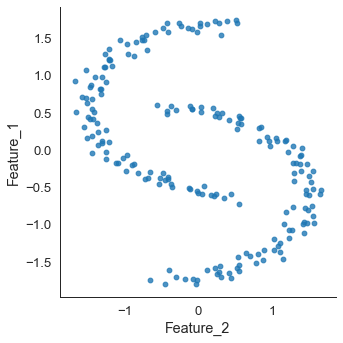
표준화를 진행한 경우 각 축의 범위가 보정된 것을 볼 수 있다.
- 표준화, 정규화에 대한 간단한 설명
https://jinyes-tistory.tistory.com/144?category=855830
2-3 dbscan
## DBSCAN
dbscan = DBSCAN(eps = 0.5, min_samples = 5, metric='euclidean')
dbscan.fit(moons_scaled)
moons_scaled['DBSCAN Cluster Labels'] = dbscan.labels_
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
hue = "DBSCAN Cluster Labels", data = moons_scaled, fit_reg = False)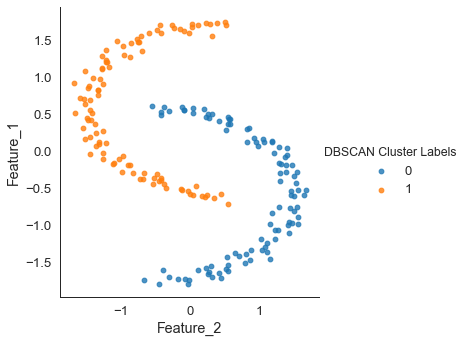
굉장히 분류가 잘된 것을 볼 수 있다. dbscan은 위와 같은 데이터에서 좋은 성능을 보인다.
2-4 k-means
## k-means
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters = 2)
kmeans.fit(moons_scaled)
moons_scaled['KMeans Cluster Labels'] = kmeans.labels_
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
hue = "KMeans Cluster Labels", data = moons_scaled, fit_reg = False)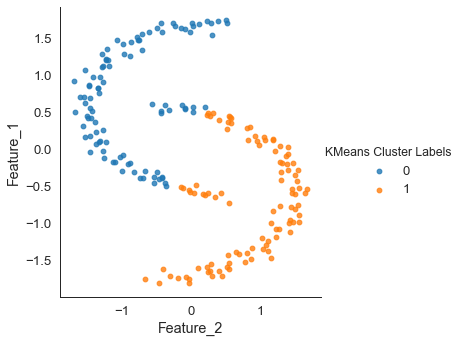
k-means의 경우 데이터의 꼬리 부분이 정확히 군집이 되지 않았다. 마치 ' / ' 의 선으로 나눈 것 같은 양상이다.
k-means는 모여있는 점들을 위주로 군집화를 하기 때문에 위와 같은 결과를 내는 것이며 따라서 해당 알고리즘이 compactness한 모델이라는 것을 확인할 수 있다.
3. iris data-set
3-1 데이터 로드
## IRIS 자료
iris = datasets.load_iris()
species = pd.DataFrame(iris.target)
species.columns = ['species']
data = pd.DataFrame(iris.data)
data.columns = ['Sepal.length','Sepal.width','Petal.length','Petal.width']
petal = data.iloc[:, 2:]
petal.head()
3-2 k-means
## k-means
kmeans = KMeans(n_clusters = 2)
kmeans.fit(petal)
petal['KMeans Cluster Labels'] = kmeans.labels_
sns.lmplot(x = 'Petal.length', y = 'Petal.width',
hue = "KMeans Cluster Labels", data = petal, fit_reg = False)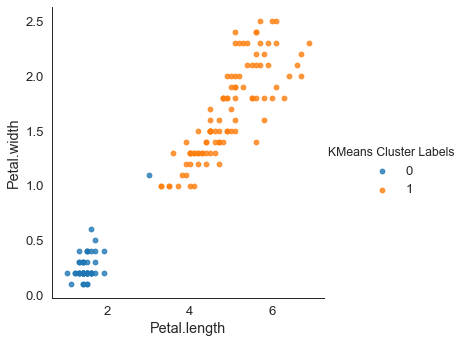
오렌지색 쪽에 데이터 하나가 이상하게 군집화된 모습을 보인다.
3-3 dbsacn
## DBSCAN
petal = data.iloc[:, 2:]
dbscan = DBSCAN(eps = 0.9, min_samples = 5, metric = 'euclidean')
dbscan.fit(petal)
petal['DBSCAN Cluster Labels'] = dbscan.labels_
sns.lmplot(x = 'Petal.length', y = 'Petal.width',
hue = "DBSCAN Cluster Labels", data = petal, fit_reg = False)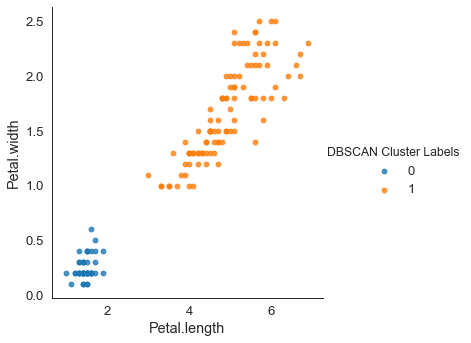
반면에 dbscan은 깔끔하게 군집화에 성공했다.
3-4 표준화
## 표준화
scaler = StandardScaler()
petal = data.iloc[:, 2:]
scaler.fit(petal)
petal_scaled = scaler.transform(petal)
petal_scaled = pd.DataFrame(petal_scaled, columns = ['Petal.length','Petal.width'])
print(petal_scaled)
3-4-1 k-means
## k-means
kmeans = KMeans(n_clusters = 2)
kmeans.fit(petal_scaled)
petal_scaled['KMeans Cluster Labels'] = kmeans.labels_
sns.lmplot(x = 'Petal.length', y = 'Petal.width',
hue = "KMeans Cluster Labels", data = petal_scaled, fit_reg = False)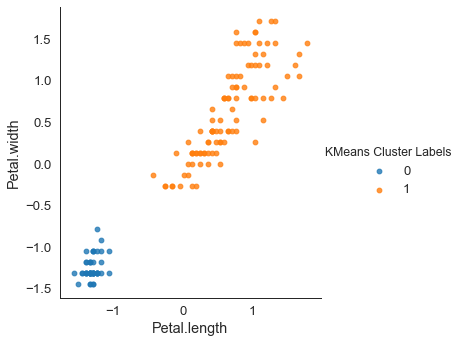
표준화를 진행한 데이터에서는 k-means도 정확히 분류가 가능하다.
데이터 전처리의 중요성을 보여주는 사례라고 할 수 있다.
3-4-2 dbscan
## DBSCAN
dbscan = DBSCAN(eps = 0.5, min_samples = 5, metric='euclidean')
# fit to input data
dbscan.fit(petal_scaled)
# get cluster assignments
petal_scaled['DBSCAN Cluster Labels'] = dbscan.labels_
sns.lmplot(x = 'Petal.length', y = 'Petal.width',
hue = "DBSCAN Cluster Labels", data = petal_scaled, fit_reg = False)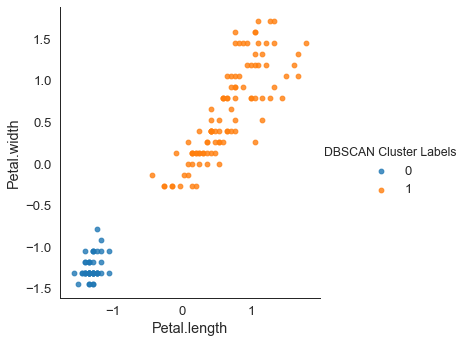
4. spectral
4-1 샘플 데이터 (blobs func)
## Spectral clustering
## 장점: k-means, dbscan에 비해 우수한 성능
## 단점: 계산량이 많다, 클러스터 개수를 결정하기 어렵다.
## blobs
df = make_blobs()
from sklearn.cluster import SpectralClustering
# 주성분 이후 k-means 사용 = assign_labels = "kmeans"
# n_init = 처음 시작할때 군집으로 보이는 개수
# n_neighbors = 인근에 평균을 기준으로 찾아냄
# affinity = 유사성, 가까운 이웃을 기준으로 찾음
clus = SpectralClustering(n_clusters = 5, random_state = 42,
assign_labels = 'kmeans', n_init = 10,
affinity = 'nearest_neighbors', n_neighbors = 10)
clus.fit(df)
df['Spectral Cluster Labels'] = clus.labels_
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
hue = "Spectral Cluster Labels", data=df, fit_reg=False)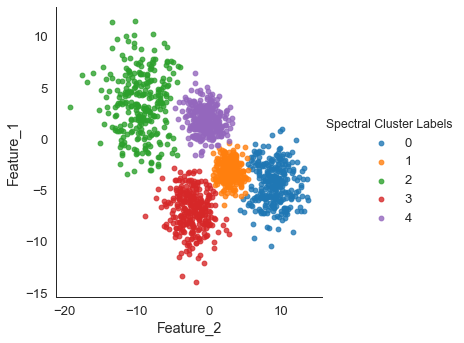
dbscan은 군집화에 실패했는데, spectral은 깔끔하게 군집화에 성공했다.
4-2 moons 데이터
## moons
df = make_moons()
clus = SpectralClustering(n_clusters = 2, random_state = 42,
assign_labels = 'kmeans', n_init = 10,
affinity = 'nearest_neighbors', n_neighbors = 10)
clus.fit(df)
df['Spectral Cluster Labels'] = clus.labels_
sns.lmplot(x = 'Feature_2', y = 'Feature_1',
hue = "Spectral Cluster Labels", data=df, fit_reg=False)
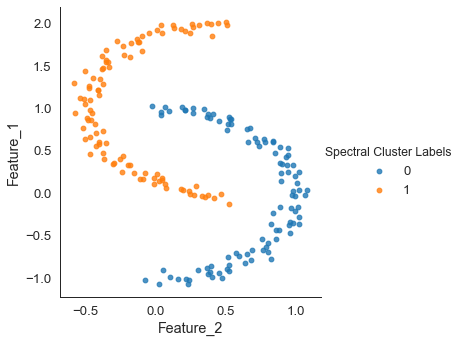
moons 데이터도 정확히 분류한다.
'Homework > DataMining' 카테고리의 다른 글
| 11. Logistic regression - deep learning (0) | 2020.06.02 |
|---|---|
| 10. OLS, SGD (0) | 2020.05.26 |
| 08. Clustering - K-means, Hierarchical (0) | 2020.05.12 |
| 07. PCA(Pincipal Component Analysis) (0) | 2020.05.05 |
| 06. Feature Selection (0) | 2020.05.01 |



